小迈带你一文看懂基因检测报告,解读基因变异命名规则
1 导读
当你拿到一份基因检测报告,面对报告中基因多种多样的字母、数字和符号的写法,是不是一时傻傻分不清楚呢?今天小迈就带领大家一起来学习一下基因变异的命名规则。
从1977年第一代DNA测序技术(Sanger法)发展至今的四十多年时间里。随着二代测序技术的发展, 越来越多与疾病发生、发展密切相关的基因突变被鉴定出来。为了将基因突变的结果更好地转化为实际临床应用,方便对检测到的变异进行准确且标准化的描述和共享。序列变异的命名必须符合精准、明确、稳定的原则,具备一定的灵活度以描述所有已知的变异类型。人类基因组变异协会(HGVS)于2000年提出了统一而通用的序列变异命名系统。2015年HGVS推出新的版本HGVS15.11,修正了原来版本的错误,去除繁琐的表述,增加了对复杂突变的命名规则。
2 人类基因组变异协会(HGVS)是什么呢?
HGVS,全称Human Genome Variation Society(人类基因组变异协会)。该协会是人类遗传学协会国际基金委(International Federation of Human Genetics Societies)和人类基因组组织(Human Genome Organization,HUGO)的附属机构。协会主要负责发现和分类包括人群分布与表型相关联的人类基因组变异,并根据方法学与信息学的发展来对数据及相关的临床变异进行更新。由该协会制定的变异命名规则被最为广泛地进行使用。目前行业中普遍应用HGVS规则对变异进行命名,统一的命名规则方便了各种各样的交流和解读。
3一起学习基因变异规则中的各项内容吧!

为了提高准确度、促进序列变异的计算机分析和描述,必须严格定义变异的基本类型。更新版将变异类型分为7类:

替换(>):一个核苷酸被另一个核苷酸取代,如:g.1318G>T,表示在基因组层面,第1318位的G被T所取代。
缺失(del):一个或多个核苷酸发生缺失,如:g.3661_3706del,表示在基因组层面,第3661位到3706位的序列发生缺失。
倒位(inv):多个核苷酸被反向互补的核苷酸取代(实质上是染色体发生了倒位),如:g.495_499inv,表示在基因组层面,第495位到499位的序列突变为反向互补的序列。
重复(dup):变异位置的3’端有一个或多个相同的核苷酸插入(实质上是参考序列发生了重复),如:g.3661_3706dup,表示在基因组层面,第3661位到3706位的序列加入45个重复的核苷酸序列。
插入(ins):一个或多个核苷酸增添到某个位置上,且这些核苷酸与该位置序列不相同(以区分dup),如:g.7339_7340insTAGG,表示在基因组层面,第7339位到7340位的序列插入了一段TAGG序列。
易位(con):变异位置的一段核苷酸被基因组中另外一区域的一段核苷酸取代,如:g.333_590con1844_2101,表示在基因组层面,第333位到590位的序列被第1844位到2101位的序列取代。
插入缺失(delins/indel):一个或多个核苷酸被另外的一个或多个核苷酸取代,且不属于替换,倒位,易位,如:g.112_117delinsTG,表示在基因组层面,第112位到117位的序列发生缺失,并插入了一段TG序列。
注意:HGVS15.11版本更新后定义更加明确,有效避免了概念混淆。例如,A>T表示置换而非倒置,倒置应包含多个核苷酸序列;一个核苷酸被一个以上的核苷酸序列替代时,不应描述为置换,应属于缺失-插入变异;重复是指上游序列的串联拷贝,当插入的位置不在原始序列的下游时,应将变异类型描述为插入。插入的序列通常是短的、新生成的,并非基因组上现有序列的拷贝。较大序列的重复插入,指的是该序列在基因组其他位置有拷贝,描述时应定义原始序列的参考序列和核苷酸范围。
DNA水平变异的命名规则
DNA水平变异的命名规则
DNA水平的变异描述根据参考序列的不同可以分为三类:基因组、cDNA和非编码DNA。使用大写字母表示核苷酸。HGVS还明确声明撤回之前推荐的使用c.IVS #和c.EX#替代内含子编码的声明,这样的描述方式容易造成混淆并且不利于软件系统的开发和识别。
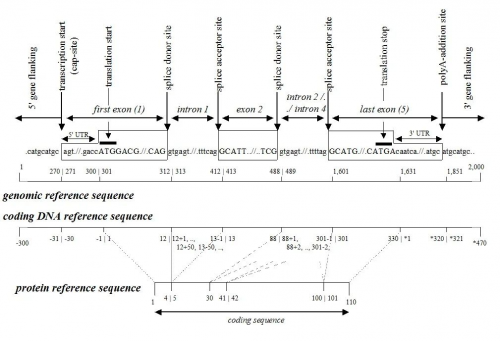
对于序列中的编号,HGVS也有严格的书写规则,不同层次的变异也有不同的编号。
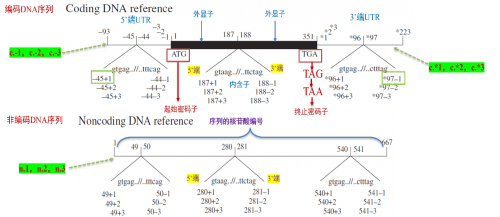
①基因组参考序列:从参考序列的第一个核苷酸开始编号,编号从“1”开始,表示为g.1,g.2,g.3,不出现“+”,“-”,“*”等符号。
②编码序列:在分子诊断领域,使用编码序列来表示变异更为普遍,因为这种表示方式能够获知变异的具体位置信息(外显子/内含子,起始密码子/终止密码子,变异的氨基酸编号)。
编码序列层面从起始密码子开始编号,编号从“1”开始,各外显子的编号是连续的,内含子及UTR不编号起始密码子上游的区域表示为c.-1,c.-2,c.-3等,终止密码子下游的区域表示为c.*1,c.*2,c.*3等。
内含子区域根据最近的外显子进行编号,靠近内含子5’端的变异,根据上游外显子的位置进行编号,如c.187+1,表示上游外显子最后一个核苷酸为187,变异为内含子5’端开始的第1个核苷酸。靠近内含子3’端的变异,根据下游外显子的位置进行编号,如c.188-1,表示下游外显子第一个核苷酸为188,变异为内含子3’端开始的第1个核苷酸。
UTR编号规则类似于内含子,如c.-123,c.*345等。而对于UTR中的内含子也有类似的规则,如c.-55+23,表示上游外显子最后一个核苷酸为-55(起始密码子上游第55位核苷酸),变异为内含子5’端开始的第23个核苷酸c.*55-23,表示下游外显子第一个核苷酸为55 (终止密码子下游第55位核苷酸),变异为内含子3’端开始的第23个核苷酸。
③非编码序列:非编码DNA的编号从参考序列的第一个核苷酸开始编号,编号从“1”开始,表示为n.1,n.2,n.3,内含子不编号,内含子区域表示方式与编码序列的方式相同。
RNA水平变异的命名规则
RNA水平变异的命名规则
RNA使用“r”标注参考序列,从编码RNA或非编码RNA参考序列的第一个核苷酸开始编号,使用小写字母表示核苷酸(a、c、g、u)。变异描述方式与DNA相同。当DNA变异影响到多个转录本时,使用“[]”标注并使用“,”连接。例如r.[76a>c,70_77del]:DNA序列上76位核苷酸由A置换为C,导致产生两个RNA产物,其中1条携带76a>c变异,另一条RNA链发生70-76位点间核苷酸的缺失(下表)。

具体举例说明:
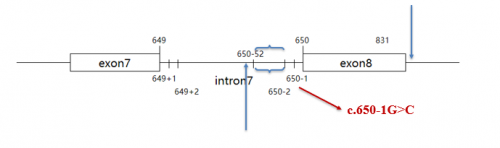
RNA层面的表述——剪接位点的突变(splicing variant)例如编码序列的变异c.650-1G>C,可能引起的剪接位点变化:①r.649_650ins[650-52_650-2;g>c],表示RNA层面,7号内含子3’端第2位到52的核酸序列插入到转录本RNA第649和650位核苷酸之间,7号内含子3’端第1位核苷酸发生了G替换为C的变异。这种变异的实质:7号内含子3’端第1位核苷酸发生变异,导致初始转录RNA剪接位点发生变化(剪接发生在7号内含子3’端第52位和第53位核苷酸之间),7号内含子的一段序列(3’端第2位到第52位核苷酸)保留在成熟转录本中。
RNA层面的表述——剪接位点的突变例如编码序列的变异c.650-1G>C,可能引起的剪接位点变化:②r.650_831del,表示RNA层面,8号外显子第650位到831位的核酸序列缺失。这种变异的实质:同①,7号内含子3’端第1位核苷酸发生变异,导致初始转录RNA剪接位点发生变化(剪接发生在8号外显子第831位和第832位核苷酸之间),最终成熟的转录本RNA中缺失了8号外显子的一段序列(第650位到第831位核苷酸)。
蛋白水平变异的命名规则
蛋白水平变异的命名规则
蛋白质“p”标注参考序列,强调蛋白水平的结果描述应标注是实验验证还是功能预测。最新版本遵循IUPAC-IUB命名方式,强烈推荐使用三个字母缩写表示氨基酸,并限制单字母描述方式在长序列变异中的应用。此外,在IUPAC-IUB中定义“X”表示任意氨基酸,为此HGVS建议使用“Ter(三字母缩写氨基酸)”或“*(单字母缩写氨基酸)”表示终止密码子,例如p.Trp123Ter或p.W123*。在移码突变的描述中增加了影响起始密码子和终止密码子变异的描述方式,对于预测的移码突变可以用两种方式展示,例如p.(Arg97fs)或p.(Arg97Profs*23)。“fsTer#”或“fs*#”使用“#”表示新的读码框以终止密码子结束,新的读码框编号从改变的氨基酸开始到终止密码子结束。同时新增变异对起始和终止密码子在N端或C端延伸的影响,使用“ext”表示,例如p.Met1ValextMet-12表示DNA序列上c.1A>G的变异导致最终蛋白产物在N端延伸了12个氨基酸,Met变异为Val。同理p.Ter110GlnextTer17表示DNA c.331T>C变异导致蛋白产物在C端多生成了17氨基酸,同时终止密码子变异为Gln。对于存在基因变异但未预测到蛋白变化的,可用“=”描述,如p.(Arg152=)(下表)。
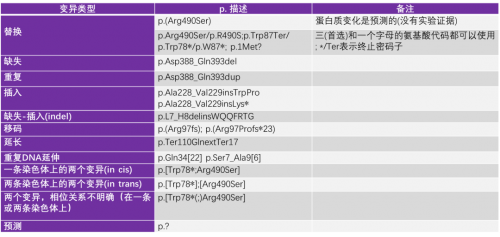
蛋白质参考序列:从第一个氨基酸开始编号,编号从“1”开始,变异的氨基酸使用通用简写符号表示(3位或1位字母简写)对于最为常见且最易看懂的蛋白质层面的变异表述,有以下几种示例:
体现突变氨基酸p.Arg490Ser或p.R490S,表示在蛋白质层面,第490位的精氨酸变异为丝氨酸BRAF V600E,BRAF蛋白第600位的缬氨酸变异为谷氨酸EGFR T790M,EGFR蛋白第790位的苏氨酸变异为甲硫氨酸。
② 体现突变导致终止或移框无义突变(nonsense,*)p.Trp78Ter或p.Trp78*,表示在蛋白质层面,第78位的色氨酸变异为终止密码子(无义突变)移框突变(frame shift,fs)p.Arg97ProfsTer23,表示在蛋白质层面,发生了移框突变,第97位的精氨酸变异为脯氨酸,且从第97位重新开始编号(从1开始),移框突变发生后第23位突变为终止密码子
③ 体现插入/缺失:p.Asp388_Gln393del,表示在蛋白质层面,第388位天冬氨酸到393位谷氨酰胺之间缺失5个氨基酸。
以上就是小迈从不同水平向大家解释了基因变异命名规则。相信随着新一代测序技术应用范围的不断扩大,会有更多的变异基因被不断发现,统一而通用的基因突变命名规则也会对科技工作者分析、解读报告信息提供便携。与此同时,迈基诺也期待着在测序技术应用范围不断扩大的基础上,能够挖掘出更多的药物作用靶点,为精准治疗提供更多的帮助。
参考文献
[1] den Dunnen et al. HGVS Recommendations for the Description of Sequence Variants: 2016 Update.Hum Mutat.2016 Jun;37(6):564-9.
[2] Hart RK et al. A Python package for parsing, validating, mapping and formatting sequence variants using HGVS nomenclature. Bioinformatics.2015 Jan;31(2):268-70.
[3] den Dunnen et al. Nomenclature for the description of human sequence variations.Hum Genet.2001 Jul;109(1):121-4.
 健康教育网
健康教育网